Descubren la primera recombinación del SARS-CoV-2 con una amplia propagación

Científicos estudiando diferentes muestras en un laoratorio / FdV
Investigadores de la Universidad de Oxford y de otras instituciones científicas de Reino Unido, México y Brasil han descubierto el primer linaje recombinante del coronavirus SARS-CoV-2 que ha tenido una amplia propagación en el tiempo y en el espacio.
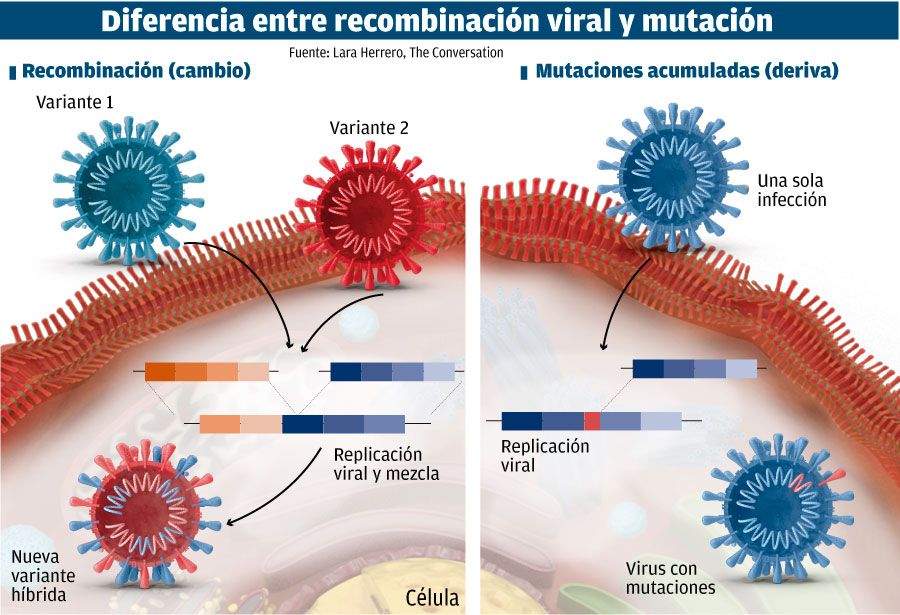
Se halló en 41 Estados de EE UU y 31 de México, así como en países de Centroamérica, y también en Europa. La recombinación se produce cuando dos partículas virales de dos linajes diferentes entran en la misma célula, intercambian segmentos genéticos y producen una nueva combinación. Es un proceso natural y ya constatado en el coronavirus que causa el COVID-19, pero hasta ahora no se había descrito un linaje recombinante tan viable y duradero.
Se trata del linaje B.1.628 y el trabajo figura como preimpresión con el título “Emergence and widespread circulation of a recombinant SARS-CoV-2 lineage in North America” (Aparición y circulación generalizada de un linaje del SARS-CoV-2 recombinante en América del Norte).
“La importancia del trabajo radica en haber encontrado un evento de recombinación que ha dado lugar a variantes viables”, explica a FARO Marina Escalera Zamudio, investigadora del departamento de Zoología de la Universidad de Oxford y científica del Consorcio Mexicano de Vigilancia Genómica. Explica que la recombinación no está asociada necesariamente a mayor transmisibilidad, virulencia o escape a vacunas o pruebas diagnósticas, pero “podría ser una fuente, porque aporta diversidad genética”. En este caso particular, los investigadores no encontraron ningún indicativo que muestre que B.1.628 presenta una mayor transmisibilidad, virulencia o alguna propiedad biológica diferente a otras poblaciones virales.
El autor principal del estudio, Bernardo Gutiérrez, que junto con Marina Escalera habló con FARO por videoconferencia, señala que hallaron “más de 1.000 genomas, principalmente distribuidos entre México y Estados Unidos, pero hay casos en otros países de Centroamérica y en otros continentes”.
“El punto principal del estudio es el tiempo que ha durado este linaje recombinante”

Bernardo Gutiérrez- Científico Universidad de Oxford / Rafa López
Gutiérrez, perteneciente también al departamento de Zoología de la Universidad de Oxford y al Consorcio Mexicano de Vigilancia Genómica, explica que el trabajo se realizó con cuatro linajes del virus: B.1.627, B.1.628, B.1.631 y B.1.634. Uno de los autores del artículo detectó que uno de estos cuatro linajes tenía similitudes genéticas mayores de las que cabría esperar entre todos los linajes surgidos en estos casi dos años de evolución viral. “Logramos identificar dos linajes que son los parentales y el linaje recombinante que surge, que ha heredado una porción del genoma de uno de los dos que estábamos estudiando y otra porción de otro linaje que estudiábamos”. “Tuvimos que hacer una serie de análisis complejos para tratar de determinarlo –interviene Marina Escalera–. La diversidad del virus es muy baja, es tan parecido que encontrar estos eventos resulta difícil. Es una posible explicación por la que no hemos podido encontrar más recombinantes hasta ahora”.
El otro linaje recombinante del SARS-CoV-2 descrito con detalle fue el llamado XA, producto de la recombinación entre la variante alfa o británica (B.1.1.7) con la B.1.177 en una zona concreta de Inglaterra a finales de 2020. El estudio, cuyo autor principal es Ben Jackson, de la Universidad de Edimburgo, fue publicado en la revista “Cell” el pasado verano. “Fue algo que pudo identificarse por la gran cantidad de secuencias genómicas que se producen en Inglaterra –apunta Gutiérrez–. Fue un linaje muy pequeño, se identificaron 47 casos asociados”. En este trabajo en preimpresión publicado en el repositorio medRxiv, los investigadores, que han identificado más de un millar de genomas, proponen que esta variante B.1.628 sea nombrada como el linaje recombinante XB en la nomenclatura Pango, siglas en inglés de “asignación filogenética de linajes de brotes globales”. “El punto principal del estudio es el tiempo que ha durado este linaje recombinante, ha logrado propagarse mucho más que el linaje identificado en Inglaterra”, subraya el científico.
Actualmente el linaje B.1.628, o XB, está decayendo y se encamina a la extinción. Delta se impone sobre todos los linajes que van surgiendo. Sin embargo, como ha señalado en Twitter otro de los autores, Oliver G. Pybus, del Departamento de Zoología de Oxford y el Royal Veterinary College de Londres, “la recombinación como fuente de diversidad genética de virus nuevos y viables debe tomarse en serio”.
Ya en el estudio sobre el linaje XA británico, los autores señalaban que “un escenario preocupante desde una perspectiva epidemiológica es el potencial de recombinación para combinar, en el mismo genoma, mutaciones que pueden conferir propiedades de escape inmunológico con aquellas que pueden mejorar la transmisibilidad”. Bernardo Gutiérrez remarca que esta “es una hipótesis, pero no es algo que hayamos visto hasta ahora”.
Otra posibilidad es la recombinación en animales. La viróloga Margarita del Val declaró recientemente a “El Mundo” que la manera en que el SARS-CoV-2 puede crear “una nueva pandemia” sería que “el patógeno pasase a un animal y, modificado, volviese a los humanos”. Algunos científicos han sugerido que una parte de la proteína de espícula del SARS-CoV-2, la que interactúa directamente con el receptor ACE2 que el virus usa para entrar en las células, procedía de la recombinación con coronavirus de pangolín.
“La diversidad del virus es muy baja, es tan parecido que encontrar estos eventos resulta difícil”

Marina Escalera - Científica Univ. de oxford / Rafa López
Marina Escalera Zamudio recuerda que animales como los visones son reservorios del virus, y que por ello se extrema el control de las granjas. “Cuantas más poblaciones pueda infectar esto puede dar lugar a una mayor variabilidad y hacer que el virus sea propenso a cambios”, advierte, pero matiza que “antes de entrar en pánico hay que continuar con las medidas de prevención y contención, como la vacunación, porque eso es lo que va a reducir la posibilidad de que estos eventos se distribuyan”.
“Es lo esperado, lo que hacen los virus: evolucionar”, dice sobre los hallazgos de este estudio Sandra López León, médico con doctorado en Epidemiología y Genética. “Lo interesante es que es la primera vez que seguimos la evolución de un virus tan de cerca. La ventaja es que podremos saber cuándo las vacunas y las pruebas diagnósticas dejarán de servir para actualizarlas”, apunta.
Los virus de ARN, como el SARS-CoV-2, pueden experimentar dos formas de recombinación: la recombinación de ARN, que en principio puede ocurrir en cualquier tipo de virus de ARN, y el reagrupamiento, que está restringido a aquellos virus con genomas segmentados. Este segundo caso es el del virus de la gripe, el “campeón” de las mutaciones. Como explica Carl Zimmer en su libro “Un planeta de virus”, los genes del virus de la gripe están almacenados en 8 segmentos diferentes. Cuando una célula huésped empieza a fabricar los segmentos de dos virus diferentes a la vez, a veces se mezclan. La nueva descendencia recoge material genético de ambos virus, en una mezcla conocida como reagrupamiento. El reagrupamiento permite que los virus de la gripe mezclen genes en nuevas combinaciones propias, que pueden permitirles evadir nuestro sistema inmunológico y propagarse más rápidamente de persona a persona.
La recombinación es una forma de eludir el llamado “trinquete de Muller”. En genética evolutiva se denomina así al proceso por el cual, en ausencia de recombinación (especialmente en una población asexual), se produce una acumulación de mutaciones deletéreas, que en los animales aumentan la predisposición a enfermedades. En los virus, la acumulación de estas mutaciones puede llevar a su extinción. El biólogo y genetista estadounidense Hermann J. Muller (1890-1967) propuso este mecanismo como una de las razones por las que la reproducción sexual puede verse favorecida en la naturaleza frente a la reproducción asexual, ya que los organismos sexuales se benefician de la recombinación y la consiguiente eliminación de mutaciones deletéreas.
Suscríbete para seguir leyendo
- Las amas de casa ya pueden solicitar la nueva pensión de 500 euros: solo 2 requisitos
- El sencillo truco casero para recuperar el blanco de los electrodoméstico que se han vuelto amarillentos
- Una pleamar que se “come” las playas
- La mezcla definitiva para limpiar las juntas del suelo que están negras de una sola pasada
- Sufre un infarto mientras trabaja en un centro de salud de Vigo y le niegan que sea accidente laboral
- Mucho cuidado si te llaman de este número fijo: conocen tu compañía telefónica y te engañan para quedarse con tu dinero
- Vecinos alertan de que tres perros y un gato llevan meses encerrados en una casa con las persianas bajas
- Terelu responde tras la separación de Alejandra Rubio: 'Se nota